Jest pan członkiem zespołu, który zajmuje się odkrywaniem peptydów, które działają przeciw różnym drobnoustrojom. W skład zespołu wchodzą naukowcy z różnych dyscyplin. Robicie to z wykorzystaniem sztucznej inteligencji – za pomocą głębokiego modelu generatywnego HydrAMP. Zacznijmy od początku: czym są peptydy rodziny AMP, którymi się zajmujecie?
Peptydy antydrobnoustrojowe – mówiąc w wielkim skrócie – należą do rodziny bardzo krótkich białek. Geneza ich odkrycia jest ciekawa: pochodzą od żab. A żaby – jak wiadomo – żyją w środowisku obfitującym w bakterie i przez to muszą mieć przed nimi naturalną obronę. Tę obronę stanowią właśnie peptydy antydrobnoustrojowe. Klasa peptydów obecnych u żab ma bardzo interesujący mechanizm działania. Najczęściej polega on na tym, że mają one – z jednej strony – zdolność wczepiania się w błonę komórkową drobnoustroju, ale z drugiej strony jednocześnie chciałyby się od niej oddalić. Można je więc porównać do odrywającego się rzepu: ten rzep chce wniknąć do bakterii, rozedrzeć błonę komórkową, ale druga strona chce się od tej bakterii oderwać. W ten sposób błona komórkowa bakterii jest rozrywana, przez co dochodzi do śmierci bakterii.
Peptydy – jeśli powstaną takie leki – mogą więc stanowić wsparcie czy też uzupełnienie dla antybiotyków. Zapewne z czasem pojawi się oporność również na peptydy, tak jak się to stało w przypadku antybiotyków…
Szansa na to, że bakterie będą mogły stać się oporne na peptydy jest mała – właśnie z uwagi na mechanizm działania peptydów. Peptydy niszczą błonę komórkową bakterii. Tak więc pojawienie się oporności bakterii na peptydy wymagałoby modyfikacji jej błony komórkowej, a to jest ewolucyjnie bardzo trudne. Bakteriom będzie trudniej niż w przypadku antybiotyków dopasować się do takiego mechanizmu działania peptydów.
Antybiotykooporność staje się coraz większym problemem…
Rzeczywiście, jak się szacuje w 2050 r. oporność na antybiotyki będzie przyczyną większej liczby zgonów niż choroby onkologiczne.
Dlaczego tak może się stać?
Stosujemy zbyt dużo antybiotyków, m.in. w szpitalach. To właśnie szpitale stają się wylęgarnią bakterii antybiotykoopornych, bakterie zyskują tam naturalne środowisko do pojawienia się mechanizmu oporności. Drobnoustroje w naturalny sposób ewoluują i modyfikują się genetycznie. Może tak się zdarzyć, że jedna z modyfikacji spowoduje, że dana bakteria nabędzie oporność na dany antybiotyk. A potem będzie się rozprzestrzeniać, zarażając kolejnych pacjentów. Im więcej będzie podawanych antybiotyków, tam większa szansa, że jakaś bakteria stanie się oporna. A jeśli nie mamy leków na daną klasę bakterii, to te drobnoustroje zaczynają siać spustoszenie wśród pacjentów w szpitalach. Jak wiadomo, w szpitalach przebywają ludzie, których układ odpornościowy jest osłabiony. A więc te bakterie drastycznie obniżają naszą szansę na wyzdrowienie.
Potwierdzają to badania, np. przeprowadzone na Northwestern University w Chicago podczas pandemii COVID-19. Pokazały, że w szpitalach wielu pacjentów chorych na COVID było zakażonych bakteriami antybiotykoopornymi i często umierali nie tyle z powodu COVID, ile właśnie z powodu zakażenia tymi bakteriami.
Obecnie z bardzo wielu powodów rozwój nowych klas antybiotyków się zatrzymał. Większość firm farmaceutycznych nie podejmuje tego wyzwania nie tylko z powodu trudności badań naukowych, ale także kosztów rozwoju tego rodzaju antybiotyków oraz stosunkowo niewielkiego zwrotu z inwestycji. W ten sposób powstało pole do popisu dla środowiska akademickiego i… peptydów.
Czym jest model generatywny, za którego pomocą poszukiwaliście peptydów?
Najprościej można to opisać w ten sposób: dostępna jest duża liczba przykładów i chcemy za pomocą sztucznej inteligencji wygenerować nowe – np. obrazy w określonym stylu lub teksty na zadany temat. Tak samo jest, gdy mamy dostęp do ogromnej ilości peptydów i tylko część z nich jest dla nas interesująca do rozwiązania danego problemu. I tu właśnie wchodzą algorytmy sztucznej inteligencji, które mają na celu wygenerowanie nowych obiektów.
W ostatnim czasie nastąpił olbrzymi rozwój tych metod, przede wszystkim, jeśli chodzi o generowanie obrazów czy tekstów. Jednak w środowiskach biologicznych, chemicznych te metody nie radzą sobie tak dobrze jak w przypadku obrazu czy tekstu. Wynika to przede wszystkim z tego, że w naukach przyrodniczych są wykorzystywane zupełnie inne dane, jest inny paradygmat pracy.

Jaka jest pańska rola w zespole prof. Ewy Szczurek, który odkrywa peptydy? Czym się Pan w nim zajmuje?
Jestem matematykiem, zajmuję się sieciami neuronowymi oraz analizą danych. Dla mnie peptyd to sekwencja, mówiąc obrazowo – napis. Mamy litery, czyli aminokwasy, z tych liter układamy napisy, czyli peptydy. Przyjęliśmy, że w naszej pracy będziemy generować napisy będące zbiorami aminokwasów, nie dłuższe niż 26 liter.
Peptydów krótkich, tych literek, jest dużo?
Samych krótkich peptydów jest bardzo, bardzo dużo (co najmniej setki tysięcy). Po odpowiednim przygotowaniu danych okazało się, że jest co najmniej 11 tys. peptydów, o których wiemy, że są aktywne, czyli „zabijają” bakterie. Nam udało nam się wyodrębnić mniejszy podzbiór: kilka tysięcy peptydów bardzo aktywnych. Takich, których wcale nie potrzeba dużo w okolicach bakterii, żeby te „zabić”.
Jak zabraliście się do poszukiwania tych peptydów, które potencjalnie nadawałyby się na leki przeciwdrobnoustrojowe?
Jak mówiłem, z technicznego punktu widzenia każdy z peptydów jest sekwencją aminokwasów, napisem złożonym z liter; ale nie każdy taki napis jest dobrym peptydem. Tak jak w języku – każdy napis jest sekwencją liter, ale nie każda sekwencja liter ma jakiś sens.
Skonstruowaliśmy model, który bierze te napisy oraz buduje ich mapę. Unikatowe w naszym rozwiązaniu jest właśnie to, że my skonstruowaliśmy mapę wszystkich peptydów – zarówno aktywnych, jak nieaktywnych, bo w większości modeli, jakie istnieją, buduje się mapę tylko tych kilkunastu tysięcy aktywnych peptydów. Z tego powodu ogromne obszary kandydatów nie są eksplorowane, bo aby można było je przeszukiwać, ktoś musiałby je wcześniej znaleźć.
Udało nam się w bardzo efektywny sposób zamodelować język tych peptydów. Wszystkie peptydy, które zamodelowaliśmy, „miały sens”, zostały wysyntetyzowane. Czyli nasza mapa peptydów faktycznie nie pokazywała tych sekwencji, które nie miały sensu.
Powstała mapa peptydów. Co działo się dalej?
W drugim kroku w celu zidentyfikowania tych nowych peptydów wykorzystywaliśmy mapę. Interesowały nas peptydy, które same w sobie są aktywne bądź mają pewne własności. Najpierw znajdowaliśmy miejsce danego peptydu na naszej mapie, lokalizowaliśmy go. Następnie przeszukiwaliśmy otoczenie tego peptydu, czyli wykorzystywaliśmy mapę do poszukiwania wokół naszego peptydu „skarbów”, czyli bardzo aktywnych peptydów, które mogłyby być kandydatami na nowe leki.
W ten sposób osiągnęliśmy z naszej perspektywy najbardziej istotny sukces – mieliśmy nieaktywny peptyd i zamieniliśmy go w peptyd, który jest aktywny – bo znajdowaliśmy sześć jego modyfikacji, z których wszystkie były bardzo aktywne.
To otworzyło nam drogę do poszukiwania peptydów o takich własnościach, których wcześniej nie mieliśmy okazji znaleźć, bo byliśmy ograniczeni do przeszukiwania peptydów podobnych wyłącznie do tych znanych i działających.
Co dalej działo się z kandydatami? Dalej ich odsiewaliście?
Ta maszyna matematyczna, ta mapa generowała ogromną liczbę kandydatów. Synteza peptydów jest droga, więc musieliśmy wybrać spośród tych wszystkich kandydatów takich, którzy rokują najlepiej. Znowu zrobiliśmy to w kilku krokach, korzystając z ogromnej wiedzy biologicznej w naszym zespole, szczególnie mgr Pauliny Szymczak.
Najpierw wyrzuciliśmy tych kandydatów, którzy mogliby sprawić problem. W kolejnym kroku poddaliśmy te peptydy filtracji, uwzględniając modele innych naukowców dotyczące przewidywania ich aktywności. W trzecim kroku stworzyliśmy ogromy system rankingowy, żeby wybrać absolutnie najlepszych kandydatów, bo eksplorując tę mapę byliśmy w stanie wygenerować ich nawet 60 tys. Tak więc podobnie jak Google sortuje swoje wyniki, stworzyliśmy system, który porządkował nam peptydy od tych najbardziej prawdopodobnych do bycia bardzo aktywnymi do tych mniej prawdopodobnych.
W ostatnim kroku zastosowaliśmy wyniki z innego laboratorium działającego na Uniwersytecie Warszawskim – z laboratorium Piotra Setnego, który rozwija symulacje molekularne właśnie służące do oceny, czy dany peptyd wejdzie w błonę komórkową, czy będzie ją perforował, czy też nie. Tu chciałem serdecznie podziękować firmie NVIDIA. Ponieważ jedna symulacja trwa kilka dni, a ocena kilkuset naszych kandydatów trwałaby miesiące – swój czas poświęciła grupa programistów, żeby te symulacje przyspieszyć.
W rezultacie wyselekcjonowaliśmy grupę ok. 30 peptydów, które wysłaliśmy do ewaluacji w laboratorium prof. Wojciecha Kamysza w Gdańsku. Tam te peptydy zostały zsyntetyzowane i testowane na prawdziwych bakteriach. Te wyniki dały nam ostateczne potwierdzenie, że peptydy są aktywne i mają zdolność zabijania bakterii. Siedem peptydów ma te zdolności bardzo, bardzo obiecujące.
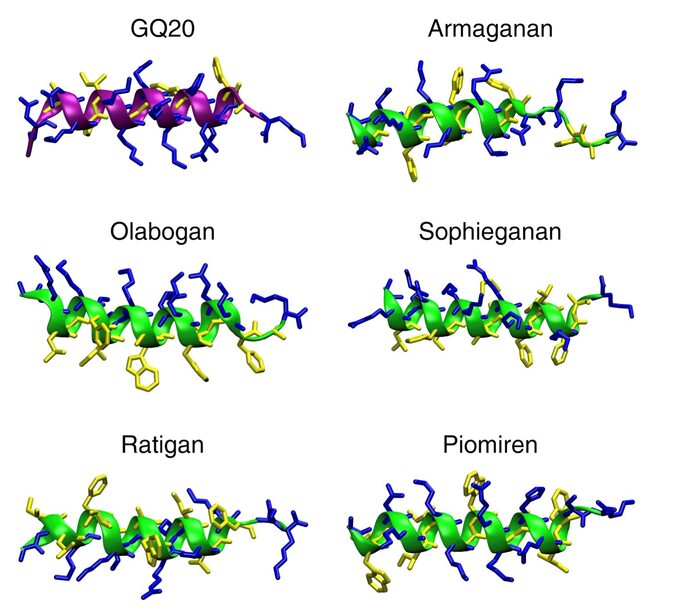
Nadaliście im nawet nazwy…
Tak, ja nazwałem jeden na część mojej ukochanej cioci, Zofii, dzięki której zostałem naukowcem. Kupowała mi encyklopedie, klocki Lego, które składałem. Dziś też „składam klocki” – no może trochę bardziej skomplikowane.
Czyli nie metoda żmudnych poszukiwań w laboratoriach, prób i błędów, ale teraz matematyka pomaga w wynajdywaniu nowych leków. Co dalej z tym pomysłem, jaka jest szansą na badania kliniczne, komercjalizację?
Nasza metoda to tylko pokaz tego, co można zrobić. Proponujemy ją wykorzystać do wygenerowania znacznie większej liczby kandydatów. Chcielibyśmy, żeby te peptydy były tak bardzo aktywne, by można było ich podawać stosunkowo niewiele. Żeby można było nimi leczyć pacjentów, konieczne jest również sprawdzenie ich toksyczności, co również testowaliśmy. Sprawdzaliśmy, czy peptydy zabijają tylko bakterie, a nie komórki pacjentów.
Trzeba podkreślić, że leków w tej kategorii jeszcze nie ma. Podanie naszego leku bakteriom jest bardzo proste: możemy dodać nasz lek do szalki i sprawdzić, czy bakterie umierają, czy nie, ale podanie tych leków pacjentom to zupełnie inna sprawa. Jest to o wiele bardziej skomplikowane. My zajmujemy się pierwszym krokiem – wyłonieniem kandydatów. Wykonanie dalszych krokówjest niemożliwe bez pomocy partnerów z firm farmaceutycznych. Jeżeli uda nam się dostarczyć odpowiednich kandydatów, postaramy się zainteresować firmy farmaceutyczne i będziemy w stanie stworzyć nowe leki.
Widzi pan szansę na to, aby tego rodzaju modele były wykorzystywane przy projektowania innych nowych leków?
Tak. Jestem osobą, która patrzy na ten problem z perspektywy matematycznej, modelowania matematycznego. To, czego potrzebowaliśmy, żeby ten model konstruował aktywne peptydy, to przede wszystkim informacji, czy są one aktywne, czy nie. Nasz model został skonstruowany, aby peptydy były aktywne przeciwko jednemu szczepowi bakterii, natomiast obecnie pracujemy nad rozszerzeniem tego modelu na wiele szczepów bakterii.
Mogę sobie łatwo wyobrazić zastosowanie tego modelu do uzyskania informacji, czy dany peptyd będzie np. zabijał komórki rakowe albo leczył inne choroby. Z perspektywy modelu matematycznego wygląda to dokładnie tak samo. Matematyka daje nam tak abstrakcyjny opis, a kolejne zastosowania są proste do wchłonięcia przez ten model.
Byłem autorem matematycznym metod sztucznej inteligencji, ale sukces naszego algorytmu miał wielu ojców, chciałbym podziękować całemu zespołowi.
Polska nauka
dla rozwoju medycyny i zdrowia Polaków
Przeczytaj inne artykuły poświęcone polskiej nauce
Projekt finansowany ze środków budżetu państwa, przyznanych przez Ministra Nauki w ramach Programu Społeczna Odpowiedzialność Nauki
Dalsze rozpowszechnianie artykułu tylko za zgodą wydawcy tygodnika Wprost.
Regulamin i warunki licencjonowania materiałów prasowych.
